
Figura 1 – O uso da inteligência artificial escancarou novas perspectivas e possibilidades no estudo das estruturas 3D e na formação de complexos de proteínas. A revista Science elegeu o assunto como um dos maiores avanços científicos do ano de 2021 [9]. (Crédito: Spencer Phillips/EMBL-EBI, Solving the protein structure puzzle EMBL)
Por: Prof. Roberto N. Onody *
O ano de 2021 foi disruptivo para a biologia estrutural de proteínas. Foi o ano em que a inteligência artificial obteve resultados inimagináveis há 50 anos atrás (Figura 1). Além de colocar o problema do enovelamento de proteínas em um novo patamar, a inteligência artificial abriu caminho para que, com orçamentos mais modestos, cientistas e laboratórios do mundo todo possam, agora, desenvolver e aprofundar suas pesquisas.
As proteínas são os blocos de construção e manutenção da vida como a conhecemos. Não importa qual seja o seu domínio na evolução celular – Bacteria, Archaea ou Eukarya, as proteínas são fundamentais e estão sempre presentes.
Proteínas são grandes cadeias lineares de aminoácidos (quando essas cadeias são pequenas, são chamadas de peptídeos). Os aminoácidos são compostos orgânicos que contêm carbono, oxigênio, hidrogênio e nitrogênio. Ao se ligarem entre si, os aminoácidos adquirem (num piscar de olhos) uma estrutura tridimensional que, juntamente com a sequência dos diferentes tipos de aminoácidos conectados, definirão a proteína resultante e a sua funcionalidade. Esse processo é chamado de enovelamento da proteína (“protein folding”).
Em um minuto, o ser humano produz cerca de 120.000 proteínas.
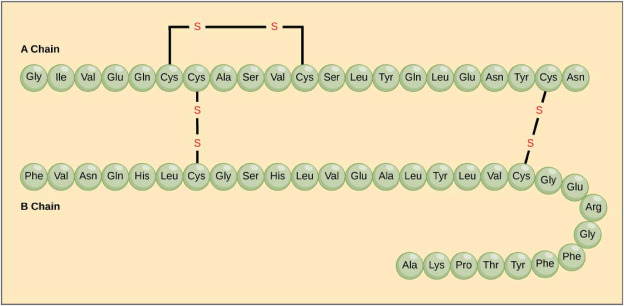
Figura 2 – Descoberta em 1921, a insulina é um hormônio sintetizado no pâncreas. É fundamental no metabolismo dos carboidratos, principalmente da glicose. É formada por 2 cadeias de peptídeos (uma com 21 aminoácidos e a outra com 30 aminoácidos) ligadas entre si por átomos de enxofre. Sua fórmula contém 788 átomos – C257 H383 N65 O77 S6 (Crédito: ref. [1])
É bem conhecido o mecanismo de síntese de proteínas por expressão gênica.
Em eucariotos, um gene do DNA (que está no núcleo da célula) é copiado (transcrito) para um RNA. Este, por sua vez, passa por um processamento bioquímico que o transforma num RNAm (RNA mensageiro). O RNAm atravessa a membrana do núcleo para o citoplasma e, no ribossomo, através dos códons e dos RNAt (RNA transportador), convoca os aminoácidos corretos e sintetiza a proteína desejada.
A expressão gênica pode sintetizar um pouco mais do que 20.000 tipos de proteínas (número estimado de genes humanos). Com a regulação pós-transcrição (“alternative splicing”), o número total de tipos de proteínas pode chegar próximo de 200.000. Estima-se que, num volume muito pequeno de 10 – 15 litros (igual a 1 micrômetro cúbico) de uma célula, existam de 2 a 4 milhões de proteínas. E olhe que o número total de células existentes no corpo humano é estimado em 30 trilhões!
A primeira proteína que teve sua sequência de aminoácidos corretamente determinada, foi a insulina (por F. Sanger, 1949). O fígado, os músculos e as hemácias (também conhecidas por glóbulos vermelhos ou eritrócitos) contêm 30% de proteínas. As proteínas também recebem outras denominações que dependem de sua função dentro do organismo.
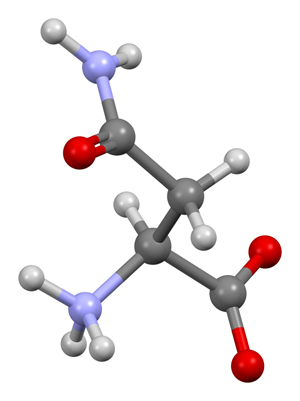
Figura 3 – Asparagina – o primeiro aminoácido descoberto. Cores: cinza escuro – Carbono; cinza claro – Hidrogênio; lilás – Nitrogênio; vermelho – Oxigênio. Fórmula: C4 H8 N2 O3 (Crédito: ref. [2])
As enzimas, por exemplo, são proteínas fundamentais com a função de catalisar reações químicas, seja na produção de novas substâncias, seja na sua degradação. Assim, a pepsina ou protease degrada proteínas em moléculas menores, a miosina atua na contração muscular, a lactase facilita a hidrólise da lactose, a DNA polimerase atua na duplicação do DNA etc. A atividade enzimática é controlada, principalmente, pela temperatura e pelo pH.
Os hormônios são proteínas que têm função regulatória das atividades fisiológicas e manutenção da homeostase. São segregados pelo nosso sistema endócrino. Entre os mais importantes podemos citar: a insulina (Figura 2), que metaboliza o açúcar; a cortisona, no combate às inflamações e os hormônios sexuais estrogênio e testosterona.
Cada órgão sintetiza seu próprio conjunto de proteínas necessárias para o seu pleno funcionamento. No entanto, para sintetizar uma proteína, o organismo tem que ter disponibilidade de todos os aminoácidos que a compõem. As plantas sintetizam todos os tipos de aminoácidos (hoje, são conhecidos mais de 500 aminoácidos), mas os animais não. Existem 20 tipos de aminoácidos nos animais, sendo que 9 deles são essenciais e não são sintetizados pelos animais. Precisamos, então, nos alimentar das plantas para obtê-los (ou de animais que delas se alimentaram). O prato brasileiro principal, feijão com arroz, contém todos os 9 aminoácidos essenciais.
Em 1806, químicos franceses encontraram, no aspargo, o primeiro aminoácido – a asparagina (Figura 3), que não é um aminoácido essencial. Na construção de uma proteína, o primeiro aminoácido incorporado é a metionina (Figura 4). É um aminoácido essencial, codificado pelos códons AUG. É encontrado em grãos, sementes, ovos, carne e peixes.
Durante muitas décadas, os experimentos para se conseguir a estrutura de uma proteína, necessitavam antes cristalizá-la para depois submetê-la a experimentos de espalhamento de raios-x. Um processo muito demorado.
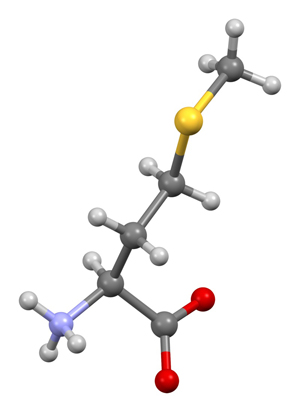
Figura 4 – Metionina – um aminoácido essencial. Cores: cinza escuro – Carbono; cinza claro – Hidrogênio; lilás – Nitrogênio; vermelho – Oxigênio; amarelo – Enxofre. Fórmula: C5 H11 N O2 S (Crédito: ref. [3])
A partir de 2001, também passou a ser utilizada a técnica de espectroscopia de ressonância magnética nuclear [4]. Ela permite calcular como os átomos estão ligados quimicamente, suas distâncias e velocidades. Em geral, as amostras são dissolvidas em água. Essa técnica, permite também determinar a dinâmica de interação de duas proteínas.
Desde sua invenção (na década de 1930) o microscópio eletrônico de transmissão muito tem contribuído ao estudo de biomoléculas e novos materiais. A preparação de amostras biológicas em baixa temperatura, o avanço na tecnologia dos detetores e de softwares, conduziram ao desenvolvimento da microscopia eletrônica criogênica [5]. Amostras biológicas rapidamente congeladas em gelo amorfo (vítreo) têm pouco dano estrutural. O sucesso dessa nova técnica levou ao prêmio Nobel de Química de 2017.
Todas as proteínas com sua estrutura tridimensional resolvida experimentalmente (por qualquer um dos três métodos descritos acima) estão contidas num repositório de livre acesso – “Protein Data Bank” [6]. São cerca de 185.000 proteínas catalogadas. Estima-se que existam pouco mais de 700.000 proteínas no corpo humano (com e sem estrutura espacial conhecida).
Como vimos, ao juntar dezenas ou centenas de aminoácidos (e de tipos de aminoácidos) para formar uma determinada proteína, ao final, devido às interações entre os seus componentes, a proteína se enovela, isto é, ela se curva e se retorce, adquirindo uma estrutura tridimensional que terá papel fundamental na funcionalidade dessa proteína (e sua eventual utilização no design de novas drogas). Há 50 anos atrás, o prêmio Nobel de bioquímica, C. Anfinsen, previu que um dia seria possível se determinar a estrutura 3D final de qualquer proteína, a partir da sequência de aminoácidos que a compõe.
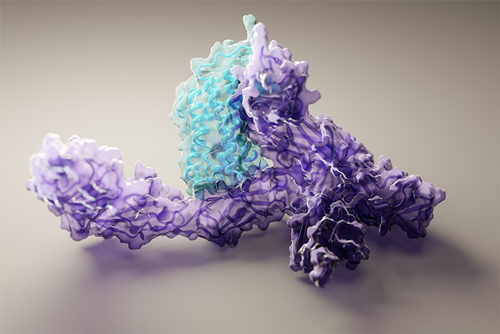
Figura 5 – A estrutura da proteína humana interleucina-12 se ligando a seu receptor (Crédito: Ian Haydon, UW Medicine Institute for Protein Design)
Dessa maneira, prever a estrutura espacial de uma proteína a partir da sua sequência de aminoácidos, se tornou um enorme desafio teórico para físicos, químicos e biólogos. Paralelamente, na década de 1990, surgiram programas computacionais que procuravam prever a estrutura de pequenas proteínas. Em 1994, foi lançada uma competição bianual chamada CASP (Critical Assessment of protein Structure Prediction). Aos competidores eram fornecidas as sequências de aminoácidos de algumas dezenas de proteínas, e os resultados desses programas computacionais eram, então, comparados com os resultados experimentais. Um porcentual de 90 % de acerto na estrutura seria considerado um sucesso.
No início, os programas mal chegavam perto do índice de 60%. Mas, em 2018, entrou na competição um programa de inteligência artificial chamado AlphaFold [7], desenvolvido pela companhia DeepMind, subsidiária da Google. Ela atingiu o índice de 80% e, em 2020, a sua segunda versão AlphaFold 2, obteve uma precisão incrível de 92,4 %! O custo computacional é bastante alto, pois utiliza 182 processadores otimizados para aprendizado de máquina. Em 2021, entrou em cena um outro programa de inteligência artificial, o RoseTTAFold [8] com os mesmos objetivos do AlphaFold, mas que demanda menos poder computacional. Ambos os programas têm seus códigos disponíveis gratuitamente na internet.
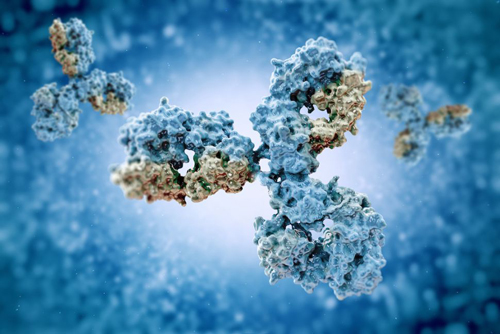
Figura 6 – Os anticorpos são proteínas em forma de Y que se ligam a proteínas estranhas ou nocivas. O fato de serem bem grandes, permite que elas se liguem à proteína alvo em vários pontos de encaixe (Crédito: Shutterstock)
Basicamente o que esses programas de inteligência artificial [10], [11] fazem é alimentar uma rede neural com uma determinada sequência de aminoácidos (de uma proteína com estrutura 3D experimentalmente conhecida) e alterar vários bilhões de parâmetros para que saída seja uma proteína compatível com aquela experimental. Repete-se esse procedimento, dando entrada para centenas de milhares de proteínas com estruturas 3D experimentalmente conhecidas. Uma nova proteína, com a sua própria sequência de aminoácidos, tem sua estrutura calculada com os valores dos bilhões de parâmetros ´treinados´ ou ´ensinados´ exaustivamente. A estrutura 3D dessa proteína vai para um banco de dados. A AlphaFold tem quase um milhão de estruturas proteicas propostas para o proteoma humano. A previsão de novas estruturas pode auxiliar no design de novas drogas (Figura 5). Juntos os dois programas revelaram mais de 5.000 complexos de interação proteína-proteína. Pesquisadores chineses mapearam a estrutura de 200 proteínas que se ligam ao DNA.
Anticorpos são proteínas grandes em forma de Y, que se conectam a proteínas prejudiciais presentes em bactérias, vírus ou células cancerígenas, sinalizando ao sistema autoimune para que destrua o invasor (Figura 6). Pelo fato de serem grandes, os anticorpos podem se conectar, simultaneamente, a vários pontos da proteína alvo (antígeno). Anticorpos produzidos artificialmente são, porém, caros e instáveis. Dessa maneira, pesquisadores se lançaram à tarefa de produzir mini proteínas, mais estáveis, mas que necessitam ser bem ´desenhadas´ para se encaixarem em determinados locais e regiões da proteína alvo. Em artigo recente publicado na revista Nature [12], cientistas utilizaram o programa RoseTTAFold para encontrar a sequência de aminoácidos de mini proteínas que se encaixaram em 12 proteínas alvo, incluindo a do SARS-CoV-2.
Claro, apesar do enorme progresso e da rapidez que os programas de inteligência artificial trouxeram à previsão estrutural das proteínas, isso não significa, stricto sensu, que o problema do enovelamento de proteínas tenha sido resolvido. Muitos de nós gostaríamos de ver soluções baseadas em primeiros princípios, ou seja, nas interações físicas e químicas.
*Físico, Professor Sênior do IFSC – USP
e-mail: onody@ifsc.usp.br
Para acessar todo o conteúdo do site “Notícias de Ciência e Tecnologia” dirija a câmera do celular para o QR Code abaixo.

Compartilhe:

(Agradecimento: ao Sr. Rui Sintra da Assessoria de Comunicação)
Referências:
[1] Insulin: A pacesetter for the shape of modern biomedical science and the Nobel Prize – ScienceDirect
[2] Ben Mill, Public Domain
https://commons.wikimedia.org/w/index.php?curid=98314887
[3] Ben Mill, Public Domain
https://commons.wikimedia.org/w/index.php?curid=98248870
[4] The way to NMR structures of proteins | Nature Structural & Molecular Biology
[6] wwPDB: Worldwide Protein Data Bank
[7] AlphaFold Protein Structure Database (ebi.ac.uk)
[8] The Rosetta Software | RosettaCommons
[9] Science’s 2021 Breakthrough of the Year: AI brings protein structures to all | Science | AAAS
[10] Teste de Turing e Inteligência Artificial – Portal IFSC (usp.br)
[11] C4AI – Centro de Inteligência Artificial (usp.br), parceria da USP, Fapesp e IBM.
[12] Design of protein binding proteins from target structure alone | Nature
Assessoria de Comunicação – IFSC/USP



